无处不在的细菌如何被检测 无处不在的细菌如何被检测 健明迪
无处不在的细菌如何被检测? 无处不在的细菌如何被检测? 健明迪
阴道微生态检测,究竟检测什么呢?
阴道微生态检测是阴道炎症诊断的抢手新词,如今很多大医院也都有了阴道微生态检测这个项目。那么阴道微生态检测究竟检测什么呢?
普通来说阴道微生态检测分为形状学检测和功用学检测。
形状学检测:包括菌群密集度、菌群多样性、优势菌、滴虫、真菌等。
功用学检测:包括PH值、过氧化氢、唾液酸苷酶、白细胞酯酶、ß-葡萄糖醛酸苷酶、乙酰氨基糖苷酶等。
阴道微生态诊断方案为形状学结合功用学检测,形状学检测为主,功用学检测为辅。若形状学检测与功用学检测结果不分歧时,目前以形状学检测为主要参考目的。
VVC和TV诊断相对比拟复杂,普统统过对白带的显微镜观察,找到病原可以诊断,但AV、BV、CV、DV则复杂得多,单纯从形状上很难找到病原,更多的是从白带的一些功用反省来确诊。
1、形 态 学 检 测
检测方法:镜检法,油镜下反省阴道菌群。
菌群密集度:
依据100倍油镜下每个视野的平均细菌数分为Ⅰ~Ⅳ级。Ⅰ级:1~10个菌/油镜;Ⅱ级:10~100个菌/油镜;Ⅲ级:100~1000个菌/油镜;Ⅳ级:1000以上/油镜。
正常为Ⅱ~Ⅲ级,Ⅳ级为菌群增殖过度。
菌群多样性:
依据100倍油镜下视野区分出的细菌菌群数目可分为Ⅰ~Ⅳ级。Ⅰ级:1~3种菌群/油镜;Ⅱ级:4~6种菌群/油镜视野;Ⅲ级:7~9种菌群/油镜;Ⅳ级:10种以上菌群/油镜。
正常为Ⅱ~Ⅲ级。
优势菌:
油镜下所见*多的微生物可以定义为优势菌, 正常应检出革兰阳性杆菌。
菌群抑制:缺少优势菌, 阴道菌群多样性≤Ⅰ级。
菌群增殖过度:优势菌为乳酸杆菌, 但菌群密集度出现增高, 为Ⅳ级。
病原微生物:
主要是经过观察真菌菌丝和 (或) 滴虫,显微镜镜检阴道分泌物中能否存在滴虫或真菌假菌丝、芽生孢子、孢子等。
①真菌检测:油镜下可发现真菌卵圆形孢子、芽生孢子或管状的假菌丝,革兰染色阳性。当镜检发现芽生孢子或假菌丝时,应报告为外阴阴道假丝酵母菌病(VVC)。
②滴虫检测:革兰染色阳性,较白细胞略大,形状不规则,内有食物泡,周边有少量的白细胞或上皮细胞碎片,发现滴虫,可诊断为滴虫阴道炎。
2、功 能 学 检 测
检测方法:检测需氧菌、厌氧菌、真菌、滴虫等的代谢产物、酶的活性及pH值。
pH值:
正常状况下由于乳酸杆菌产酸,正常阴道内的pH≤4.5,多在3.8~4.4,细菌性阴道病患者的阴道pH值普通大于4.5。
过氧化氢:
阳性代表乳酸杆菌发生的过氧化氢浓度降低,阴道内乳酸杆菌增加,阴性代表乳酸杆菌正常。
唾液酸苷酶:
这是惹起细菌性阴道病的主要厌氧菌释放的,唾液酸酐酶呈阳性,意味着厌氧菌感染。是细菌性阴道病的表现。
白细胞酯酶:
以前仅仅依据白细胞数量判别炎症,目前要看白细胞吞噬细菌的功用。阴道内有炎症时,白细胞释放白细胞酯酶,白细胞酯酶增高,化验单上就标注为阳性。白细胞酯酶阳性才代表阴道炎,而白细胞增多,白带清洁度Ⅲ~Ⅳ度并不能诊断炎症。
凝结酶、ß-葡萄糖醛酸苷酶:
这是需氧菌释放的,这两个目的之一阳性,意味着需氧菌性阴道炎。
乙酰氨基糖苷酶:
是白色念珠菌的特异性酶,一旦其水平出现降低,提示患者存在阴道内膜损伤。
阴道微生态是经过这些复杂的目的反省,*后经过软件剖析停止诊断。
3、分 子 检 测
惯例镜检法是临床诊断阴道炎病原体的常用手腕,但诊断结果会遭到诸多要素影响,如分泌物涂片质量、气温、判别规范和检测人员阅历不分歧等,影响诊断准确性,容易漏检,误判。
而微生物的代谢酶数量众多, 酶学检测的效果在于, 酶的阳性与否并不是阴道炎诊断的必要要素, 以BV为例, 大约90%的BV患者会出现唾液酸酶阳性, 但是唾液酸酶阳性的患者并不一定都是BV。因此, 酶学检验只能作为形状学检验的重要补充, 有助于我们判别感染的严重水平以及混合感染等特殊状况。
牢靠的检测技术在妇科感染疾病诊治中占有十分重要的位置,分子生物学技术的出现、精准医学时代的到来,为临床上这些诊治困难的感染性疾病带来了新曙光、在微生物研讨范围中具有里程碑式的意义。
经过对30种相关生殖道有益/致病微生物的精准基因检测,一次性更精准、片面的了解阴道微生态,对阴道菌群停止生态分型评价。其高特异性、高灵敏度是替代传统镜检/培育法的有效提高。微生物鉴定与定量检验并行,提醒生殖道感染治病机理,协助女性更精准的了解阴道菌群结构。
无处不在的细菌如何被检测?
作为微生物中一个重要的研讨范围,细菌,与我们的安康和生活亲密相关,其菌种类单一,体积庞大,结构不同。在环境范围、食品范围、临床医学等,经常需求对细菌种类、细菌数量、细菌中活性物质、细菌作为媒介分解化合物、单个活性菌体等停止快速检测和剖析。
与惯例检测技术相比,显微拉曼光谱是一种快速、无损、无惧水、微区分辨、非接触式检测的分子光谱技术,十分适宜用于微生物的检测和剖析,成像功用还可以完成细菌微区成分散布的剖析。
仪器和软件
Thermo Scientific ™ DXR™ 3系列显微拉曼技术在微生物检测和剖析范围具有共同的技术和运用优势:
先进的自动化和智能化光学控制系统
超高的检测灵敏度和空间分辨率
软件控制、实时到样品的激光功率精细调理专利技术
“一键式”操作,无需专业背景
功用弱小、界面友好的剖析软件
集成数学模型算法、导游操作的TQ Analyst建模软件
光镊-微流控联用技术完成流体中微生物的快速剖析
运用案例
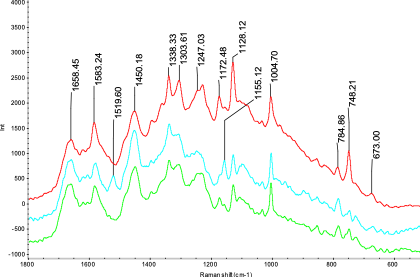
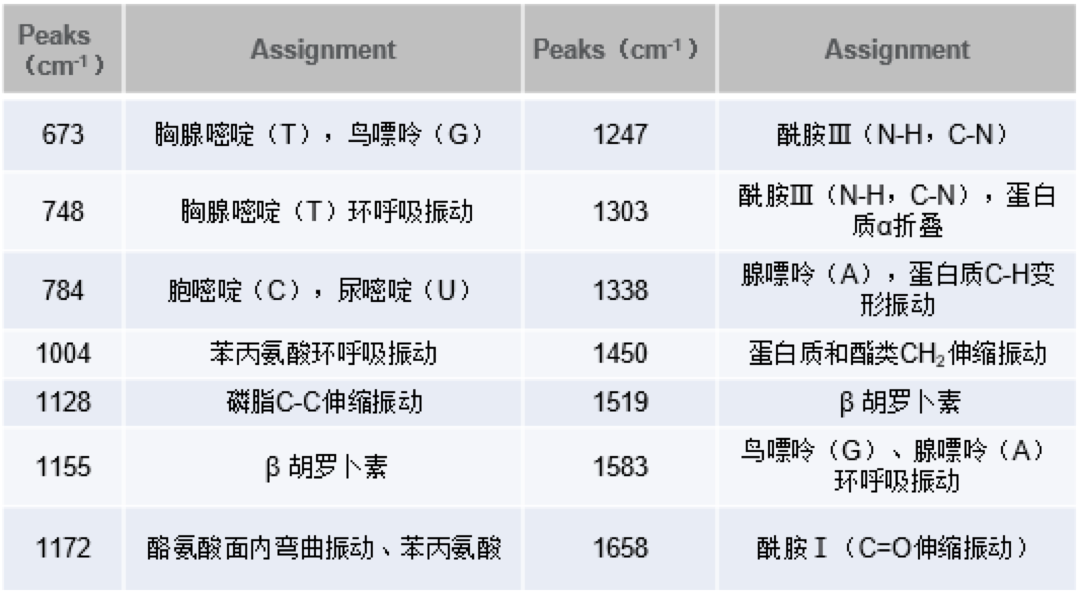
细菌结构剖析
细菌种类不同,其活性物质如氨基酸、蛋白质等表现出结构和量的差异,拉曼光谱的特征结构峰可以很好的表现活性物质和含量的差异,依据这些差异可以快速无损的停止细菌种类的鉴别和剖析。
单个细菌检测
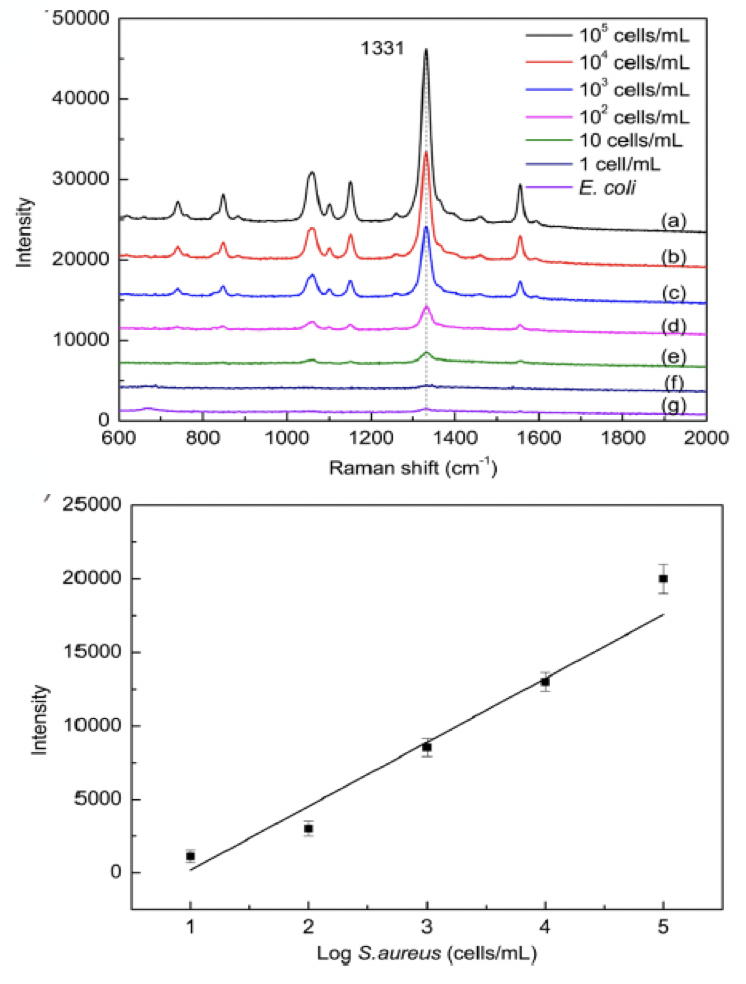
通常细菌的体积较小,信号较弱,溶液中细菌浓度较低时,很难检测到有效的化学结构信息,尤其是需求对单个细菌的化学结构停止检测和剖析。在停止分子结构剖析时,需求高灵敏度和空中间分辨率的拉曼技术。
采用超高灵敏和空中间分辨的DXRxi显微拉曼成像光谱仪,应用研讨者[1]分解的磁性富集高活性SERS传感器,经过适配子识别细菌,完成低浓度和单个金黄色葡萄球菌的检测和剖析。
单个细菌体内分解物的鉴别与剖析
酵母菌是一些单细胞真菌,其结构与初等植物的细胞一样,比拟复杂,体积小的约2-6um,大的约5-30um。在一定适宜的培育条件下,酵母菌菌体可以作为微型“反响器”,在体内分解一些目的化合物,在剖析目的化合物时,需求空中间分辨率的显微拉曼光谱仪才干实如今菌体微区的剖析,同时要求快速测试以坚持细菌的活性。而赛默飞的DXR3xi拉曼成像光谱仪因其超高灵敏度、空中间分辨率和超快速成像功用,是一款十分适宜用于这些范围的研讨。
Thermo Scientific ™DXR™系列显微拉曼光谱仪,经过蛋白质、氨基酸等生物活性物质的拉曼光谱特征性,可以轻松完成几微米甚至几百纳米大小细菌种类的检测与剖析。应用超高灵敏度和超空中间分辨的DXR3xi成像技术有助于快速研讨单个活性细菌体内物质的散布,完成微生物的活体剖析。为微生物的研讨提供了一种无损、快速、无惧水的研讨工具。
参考文献
1,Junfeng Wang et al, ACS Appl. Mater. Interfaces 2015, 7, 20919−20929
无处不在的细菌如何被检测? 无处不在的细菌如何被检测? 健明迪
SignalP+TMHMM预测微生物分泌蛋白
Secretory Protein是指在细胞内分解后,分泌到细胞外起作用的蛋白质。分泌蛋白的N 端有普通由15~30 个氨基酸组成的信号肽。信号肽是引导新分解的蛋白质向分泌通路转移的短(长度5-30个氨基酸)肽链。常指新分解多肽链中用于指点蛋白质的跨膜转移(定位)的N-末端的氨基酸序列(有时不一定在N端)。运用SignalP 注释蛋白序列能否含有信号肽结构,运用TMHMM注释蛋白序列能否含有跨膜结构,*终挑选出含有信号肽结构并且不含跨膜结构的蛋白为分泌蛋白。
软件Software
- SignalP V6.0
- SignalP 6.0 预测来自古细菌、革兰氏阳性细菌、革兰氏阴性细菌和真核生物的蛋白质中存在的信号肽predicts signal peptides and the location of their cleavage sites in proteins from Archaea, Gram-positive Bacteria,及其切割位点的位置。Gram-negative Bacteria and Eukarya.在细菌和古细菌中,SignalP 6.0 可以区分五种类型的信号肽:In Bacteria and Archaea, SignalP 6.0 can discriminate between five types of signal peptides:
- Sec/SPI:由 Sec 转座转运,并由信号肽酶 I (Lep) 切割的“规范”分泌信号肽;"Standard" secretory signal peptides transported by Sec translocon and cleaved by Signal Peptidase I (Lep).
- Sec/SPII:由 Sec 转座子运输,并由信号肽酶 II (Lsp) 切割的脂蛋白信号肽;lipoprotein signal peptides transported by the Sec translocon and cleaved by Signal Peptidase II (Lsp).
- Tat/SPI:由 Tat 转座子转运,并由信号肽酶 I (Lep) 切割的 Tat 信号肽;Tat signal peptides transported by the Tat translocon and cleaved by Signal Peptidase I (Lep).
- Tat/SPII:由 Tat 转位子转运,并由信号肽酶 II (Lsp) 切割的 Tat 脂蛋白信号肽;Tat lipoprotein signal peptides transported by Tat translocon & cleaved by Signal Peptidase II (Lsp).
- Sec/SPIII:由 Sec 转位子运输,并由信号肽酶 III (PilD/PibD) 切割的菌毛蛋白和菌毛蛋白样信号肽。Pilin & pilin-like signal peptides transported by Sec translocon & cleaved by Signal Peptidase III (PilD/PibD).
- 此外,SignalP 6.0 预测信号肽的区域。Additionally, SignalP 6.0 predicts the regions of signal peptides.依据类型,预测 n、h 和 c 区域以及其他显着特征的位置。Depending on the type, the positions of n-, h- and c-regions as well as of other distinctive features are predicted.
- TMHMM V2.0c
- 用于预测蛋白质中的跨膜螺旋。
- Python
SignalP和TMHMM关于学术用户收费,但是需求填写相关信息和邮箱,以接纳下载链接(4h有效时间)。
软件装置Installation of Softwares
装置SignalP 6.0
- 下载 访问SignalP V6.0网站,找到“Download”,填写相关信息,获取下载链接,下载失掉“signalp-6.0.fast.tar.gz”。有两个形式可以选择——“slow_sequential”和“fast"。前者runs the full model sequentially, taking the same amount of RAM as
fastbut being 6 times slower;后者uses a smaller model that approximates the performance of the full model, requiring a fraction of the resources and being significantly faste。本教程下载的是fast形式。 - 装置Installation
- 装置依赖Dependencies
- Python
- matplotlib>3.3.2
- numpy>1.19.2
- torch>1.7.0 pip install torch
- tqdm>4.46.1
- 装置SignalP 6.0 # 解紧缩装置文件 tar zxvf signalp-6.0.fast.tar.gz # 进入解压后的软件目录,在终端运转 python setup.py install # 测试装置 signalp6 --help
装置TMHMM V2.0c
- 下载 访问TMHMM V2.0c网站,找到“Download”,填写相关信息,获取下载链接,下载失掉“tmhmm-2.0c.Linux.tar.gz”。
- 装置 # 解紧缩 tar zxvf tmhmm-2.0c.Linux.tar.gz # 进入解压后的目录 cd tmhmm-2.0c # 获取以后途径,我的是“/home/liu/tools/tmhmm-2.0c/bin” pwd # 将该途径参与到系统的环境变量中,参考我之前的文章来(编辑~/.bashrc)http://liaochenlanruo.github.io/post/f6c9.html#%E6%B7%BB%E5%8A%A0%E7%8E%AF%E5%A2%83%E5%8F%98%E9%87%8F # 修正bin目录下的tmhmm和tmhmmformat.pl的首行为“#!/usr/bin/perl”
- 运转错误 运转软件时总报
Segmentation fault (core dumped)错误,暂时无解。各位可以运用其在线版。
软件用法Usage
SignalP 6.0
预测Prediction
A command takes the following form
signalp6 --fastafile /path/to/input.fasta --organism other --output_dir path/to/be/saved --format txt --mode fast
fastafile输入文件为FASTA格式的蛋白序列文件Specifies the fasta file with the sequences to be predicted.。organismis eitherotherorEukarya. SpecifyingEukaryatriggers post-processing of the SP predictions to prevent spurious results (only predicts type Sec/SPI).formatcan take the valuestxt,png,eps,all. It defines what output files are created for individual sequences.txtproduces a tabular.gfffile with the per-position predictions for each sequence.png,eps,alladditionally produce probability plots in the requested format. For larger prediction jobs, plotting will slow down the processing speed significantly.modeis eitherfast,sloworslow-sequential. Default isfast, which uses a smaller model that approximates the performance of the full model, requiring a fraction of the resources and being significantly faster.slowruns the full model in parallel, which requires more than 14GB of RAM to be available.slow-sequentialruns the full model sequentially, taking the same amount of RAM asfastbut being 6 times slower. If the specified model is not installed, SignalP will abort with an error.
输入Outputs
- output_dir/output.gff3:仅包括含有信号肽的序列信息;
- output_dir/prediction_results.txt:包括了输入文件中的一切序列(不重要);
- output_dir/region_output.gff3:包括一切的信号肽区域信息。
- n-region: The n-terminal region of the signal peptide. Reported for Sec/SPI, Sec/SPII, Tat/SPI and Tat/SPII. Labeled as N
- h-region: The center hydrophobic region of the signal peptide. Reported for Sec/SPI, Sec/SPII, Tat/SPI and Tat/SPII. Labeled as H
- c-region: The c-terminal region of the signal peptide, reported for Sec/SPI and Tat/SPI.
- Cysteine: The conserved cysteine in +1 of the cleavage site of Lipoproteins that is used for Lipidation. Labeled as c.
- Twin-arginine motif: The twin-arginine motif at the end of the n-region that is characteristic for Tat signal peptides. Labeled as R.
- Sec/SPIII: These signal peptides have no known region structure.
批处置与结果优化
脚本名:run_SignalP.pl
#!/usr/bin/perl
use strict;
use warnings;
# Author: Liu Hualin
# Date: Oct 14, 2021
open IDNOSEQ, ">IDNOSEQ.txt" || die;
my @faa = glob("*.faa");
foreach (@faa) {
$_ =~ /(.+).faa/;
my $str = $1;
my $out = $1 . ".nodesc";
my $sigseq = $1 . ".sigseq";
my $outdir = $1 . "_signalp";
open IN, $_ || die;
open OUT, ">$out" || die;
while (
chomp;
if (/^(>\S+)/) {
print OUT $1 . "\n";
}else {
print OUT $_ . "\n";
}
}
close IN;
close OUT;
my %hash = idseq($out);
system("signalp6 --fastafile $out --organism other --output_dir $outdir --format txt --mode fast");
my $gff = $outdir . "/output.gff3";
if (! -z $gff) {
open IN, "$gff" || die;
open OUT, ">$sigseq" || die;
while (
chomp;
my @lines = split /\t/;
if (exists $hash{$lines[0]}) {
print OUT ">$lines[0]\n$hash{$lines[0]}\n";
}else {
print IDNOSEQ $str . "\t" . "$lines[0]\n";
}
}
close IN;
close OUT;
}
system("rm $out");
system("mv $sigseq $outdir");
}
close IDNOSEQ;
sub idseq {
my ($fasta) = @_;
my %hash;
local $/ = ">";
open IN, $fasta || die;
while (
chomp;
my ($header, $seq) = split (/\n/, $_, 2);
$header =~ /(\S+)/;
my $id = $1;
$hash{$id} = $seq;
}
close IN;
return (%hash);
}
将run_SignalP.pl与后缀名为“.faa”的FASTA格式文件放在同一目录下,在终端中运转如下代码:
perl run_SignalP.pl
结果解读Output interpretation
*代表输入文件的名字。
- *_signalp/output.gff3:仅包括含有信号肽的序列信息;
- *_signalp/prediction_results.txt:包括了输入文件中的一切序列(不重要);
- *_signalp/region_output.gff3:包括一切的信号肽区域信息;
- *_signalp/*.sigseq:存储一切信号肽的氨基酸序列文件,可用作TMHMM的输入文件。
TMHMM
预测
离线版总是报错,找不出缘由,因此运用网页效劳器停止,输入文件为上述生成的“*_signalp/*.sigseq”,将其上传至网页版TMHMM,提交义务,等候结果即可。
结果展现
TMHMM可以输入多种格式的结果文件,详细请参考其官方说明。
在TMHMM网站提交义务
- Long output format
- Length: 蛋白序列的长度。The length of the protein sequence.
- Number of predicted TMHs:预测到的跨膜螺旋的数量。The number of predicted transmembrane helices.
- Exp number of AAs in TMHs:跨膜螺旋中氨基酸的预期数量。The expected number of amino acids intransmembrane helices. 假设此数字大于 18,则很能够是跨膜蛋白(或具有信号肽)。If this number is larger than 18 it is very likely to be a transmembrane protein (OR have a signal peptide).
- Exp number, first 60 AAs:在蛋白的前60个氨基酸中跨膜螺旋中氨基酸的预期数量。The expected number of amino acids in transmembrane helices in the first 60 amino acids of the protein.假设该数字超越几个,你应该被正告在 N 端预测的跨膜螺旋能够是一个信号肽。If it more than a few, you are warned that a predicted transmembrane helix in the N-term could be a signal peptide.
- Total prob of N-in:N端在膜的细胞质一侧的总概率。The total probability that the N-term is on the cytoplasmic side of the membrane.
- POSSIBLE N-term signal sequence:当“Exp number, first 60 AAs”大于 10 时发生的正告。A warning that is produced when "Exp number, first 60 AAs" is larger than 10.
- 蛋白F01_bin.1_00110合计436个氨基酸,有5个跨膜螺旋结构。
- 蛋白F01_bin.1_00142合计557个氨基酸,一切序列均在膜外,即该序列编码的是分泌蛋白。
- Short output format
- "len=": 蛋白序列的长度。The length of the protein sequence.
- "ExpAA=":跨膜螺旋中氨基酸的预期数量。The expected number of amino acids intransmembrane helices.假设此数字大于 18,则很能够是跨膜蛋白(或具有信号肽)。If this number is larger than 18 it is very likely to be a transmembrane protein (OR have a signal peptide).
- "First60=":在蛋白的前60个氨基酸中跨膜螺旋中氨基酸的预期数量。The expected number of amino acids in transmembrane helices in the first 60 amino acids of the protein.假设该数字超越几个,你应该被正告在 N 端预测的跨膜螺旋能够是一个信号肽。If it more than a few, you are warned that a predicted transmembrane helix in the N-term could be a signal peptide.
- "PredHel=":预测到的跨膜螺旋的数量。The number of predicted transmembrane helices by N-best.
- "Topology=":N-best 预测的拓扑结构。The topology predicted by N-best.拓扑是由跨膜螺旋的位置给出的,假设螺旋在外部,则由“i”分隔,假设螺旋在外部,则由“o”分隔。'i7-29o44-66i87-109o'意味着它从膜内末尾,在位置7到29有一个预测的TMH,30-43在膜外,然后是位置44-66的TMH。
结果汇总
经过网页版预测我们仅失掉了一个列表文件(Short output format),该文件需求自己复制网页内容粘贴到新文件中,我将其命名为*_TMHMM_SHORT.txt,并将其寄存在*_signalp目录中,该目录是由run_SignalP.pl生成的。下面我将会统计各个基因组中信号肽蛋白的总数量、分泌蛋白数量和跨膜蛋白数量到文件Statistics.txt中,并区分提取每个基因组的分泌蛋白序列到*_signalp/*.secretory.faa文件中,提取跨膜蛋白序列到*_signalp/*.membrane.faa文件中。该进程将经过tmhmm_parser.pl完成。
#!/usr/bin/perl use strict; use warnings; # Author: Liu Hualin # Date: Oct 15, 2021 open OUT, ">Statistics.txt" || die; print OUT "Strain name\tSignal peptide numbers\tSecretory protein numbers\tMembrane protein numbers\n"; my @sig = glob("*_signalp"); foreach my $sig (@sig) { $sig=~/(.+)_signalp/; my $str = $1; my $tmhmm = $sig . "/$str" . "_TMHMM_SHORT.txt"; my $fasta = $sig . "/$str" . ".sigseq"; my $secretory = $str . ".secretory.faa"; my $membrane = $str . ".membrane.faa"; open SEC, ">$secretory" || die; open MEM, ">$membrane" || die; my $out = 0; my $on = 0; my %hash = idseq($fasta); open IN, $tmhmm || die; while (
运转方法:将tmhmm_parser.pl放在*_signalp的上一级目录下,*_signalp目录中必需包括*_TMHMM_SHORT.txt文件和*.sigseq文件。在终端运转如下代码:
perl tmhmm_parser.pl
脚本获取
本文脚本见GitHub。
敬告:运用文中脚本请援用本文网址,请尊重自己的休息效果,谢谢!Notice: When you use the scripts in this article, please cite the link of this webpage. Thank you!
参考
原文链接:SignalP+TMHMM预测微生物分泌蛋白 | liaochenlanruo
转载请注明出处!